近日,福建农林大学海峡联合研究院基因组与生物技术研究中心马晓开课题组在《Briefings in Bioinformatics》期刊上发表了其团队最近开发的多功能空间转录组上下文感知深度学习框架—STMGraph,题为“STMGraph: spatial-context-aware of transcriptomes via a dual-remasked dynamic graph attention model”。STMGraph结合了双重掩码(MASK-REMASK)机制和动态图注意力模型(DGAT),旨在提高对组织中基因表达模式的空间上下文信息的感知(spatial-context-aware)能力,重点解决了原始基因表达谱混乱,无法检测微环境异质性,空间域识别精度低、多切片联合分析存在批次效应、植物组织切片中基因表达噪音高等空间组学分析和研究中的难题。文章通过系统的基准测试以及消融实验验证了模型在不同空间转录组平台的优越性以及鲁棒性。STMGraph在空间上下文信息感知方面表现优异,能够实现微环境异质性检测、空间域聚类、批次效应校正、基因表达降噪、以及具有生物学意义的新标记基因的发掘,是进行各类空间转录组研究的理想新型工具。

空间转录组技术(ST)是一种能够在组织切片中同时获取基因表达特征和细胞空间分布信息的技术。与传统的转录组测序技术相比,空间转录组技术不仅能够提供基因表达的定量信息,还能保留细胞在组织中的具体位置信息,从而更全面地揭示细胞的功能和定位关系。目前它可以根据实际实验需要测量从多细胞分辨率(10x Genomics Visium)到亚细胞分辨率(Slide-seq、Stereo-seq和STARmap)的数据 (Chen et al., 2022; Chen et al., 2021; Cho et al., 2021; Ji et al., 2020; Liu et al., 2020; Rodriques et al., 2019; Wang et al., 2018)。
组织裂解与透化所产生的基因漂移现象常常导致原始基因表达谱出现dropout-events(缺失数据引起的噪音)(Cheng, Hu, & Li, 2022; Li, Gai, Dong, Zhang, & Zhang, 2024; Wang et al., 2022; Xu et al., 2024),当前的空间域鉴定方法普遍对空间上下文感知能力弱,完全依赖原始基因表达谱的自监督训练会导致潜在嵌入引入了较多噪音。此外,背景图像的不合理使用也会进一步干扰潜在嵌入的学习过程,这使得现有的空间转录组聚类算法仍旧面临无法准确鉴别空间域的挑战 (Dong & Zhang, 2022; Hu et al., 2021; Wang et al., 2023; Xu et al., 2022; Yu et al., 2022)。
此外,低分辨率的空间转录组数据中,一个spot可能包含多个细胞,spot间的微环境差异通常需要借助其他聚类工具来衡量,这进一步影响了识别结果的准确性(Dong & Zhang, 2022)。而且,空间转录组学芯片的空间面积限制了垂直结构组织的分析,即使目前有方法实现垂直的拼接 (Zeira, Land, Strzalkowski, & Raphael, 2022),但批次效应会影响多切片联合分析的结果,降低聚类的准确性和可靠性。
本研究提出了一种基于生成图SSL的双重掩码(MASK-REMASK)动态图注意力模型(DGAT),称为STMGraph。从STMGraph生成的潜在嵌入用于微环境异质性检测、空间域聚类和批次效应校正。为了确保最相关的信息被优先考虑,研究团队使用DGAT自适应地权衡不同节点的贡献,同时记录优先级权重,以评估每个spot微环境的异质性。双视图重掩码算法高比例地掩盖ST嵌入,并通过自监督从不同角度捕获特征,以减少ST缺失的干扰。此外,DGAT通过隐式聚合不同批次区域的信息来推断潜在嵌入,有效缓解多片联合分析中的批次效应。 STMGraph在不同ST分辨率平台(10x Visium、Slide-seq、Stereo-seq和STARmap)的数据集上验证了其最佳聚类性能。总之,与现有的先进工具相比,STMGraph是一种新的模型,具有多功能性和多平台兼容性。
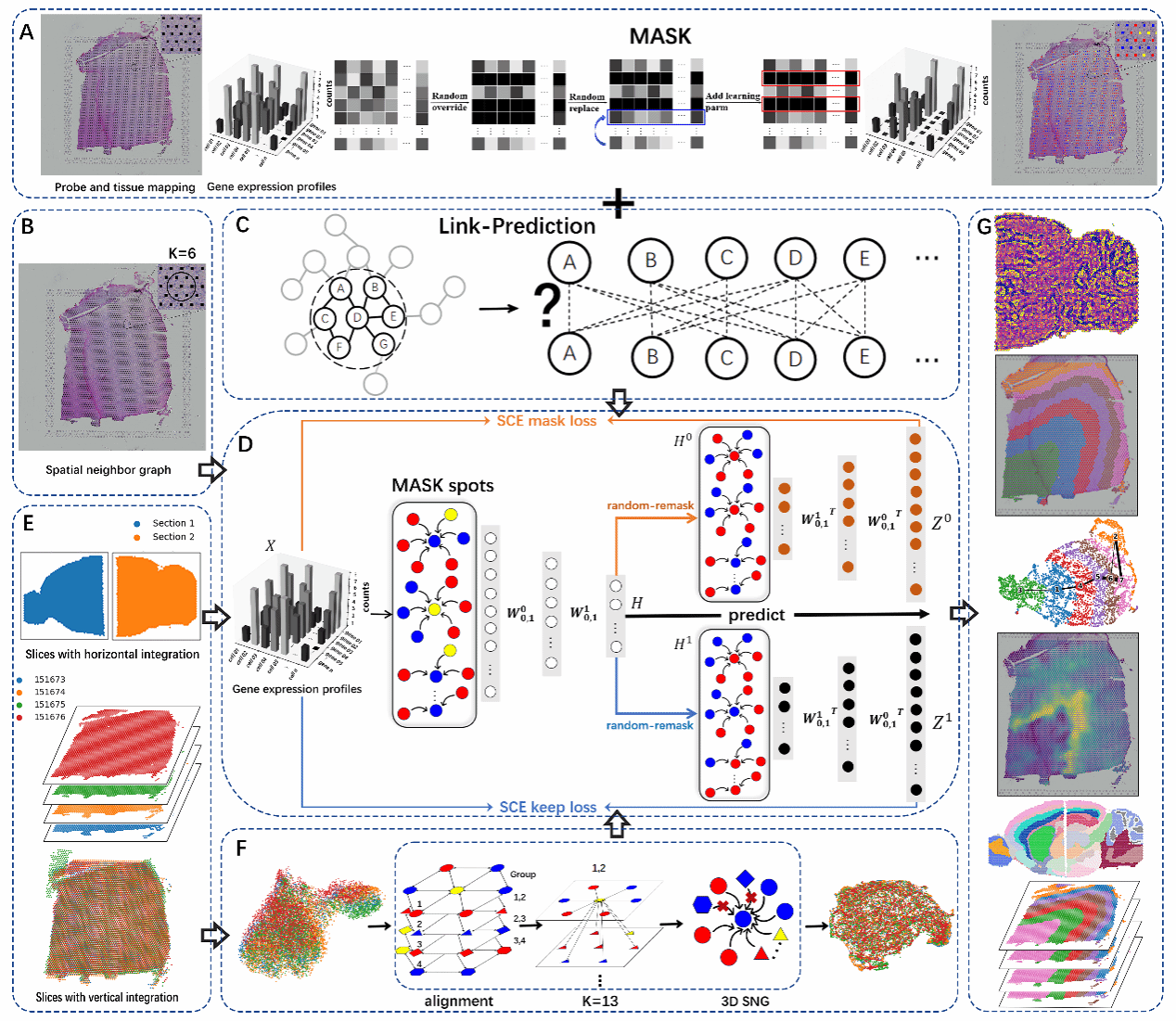
图1 STMGraph概述
STMGraph首先利用动态图注意模型(dynamic graph attention model, DGAT)对相邻ST上下文信息进行聚合,降低噪声邻域对ST上下文感知的干扰,提高潜在嵌入的信噪比。其次,双重掩码减少了输入ST特征的过拟合,并在双解码视图预测具有更多ST目标信息的潜在表示。最后,利用尺度余弦误差(SCE)作为损失函数,通过自监督对潜在嵌入进行校正。采用高比例随机掩码策略优化图结构中的特征传播过程(图1A)。它有助于揭示组织内不同区域的特征和功能,为理解微环境相互作用,特别是相邻spot之间的信号传导提供线索。该框架包括三个模块:(1)微环境异构相似性检测(图1C和G),(2)空间域聚类(图1D和G),(3)批效应校正(图1E和F)。
研究团队使用两组10×小鼠脑部切片数据,比较动态图注意机制(DGAT)和静态图注意机制(GAT)检测组织微环境中相邻点异质相似性的能力。STAGATE中静态注意力的使用阻碍了其区分相邻点的贡献的能力,从而最终阻碍了两个小鼠脑区域微观点之间建立关系。结果表明,在这种复杂的场景下,DGAT的空间-上下文感知比GAT更准确,使STMGraph能够在不进行预聚类的情况下考虑相邻点之间的异质相似性(图 2)。
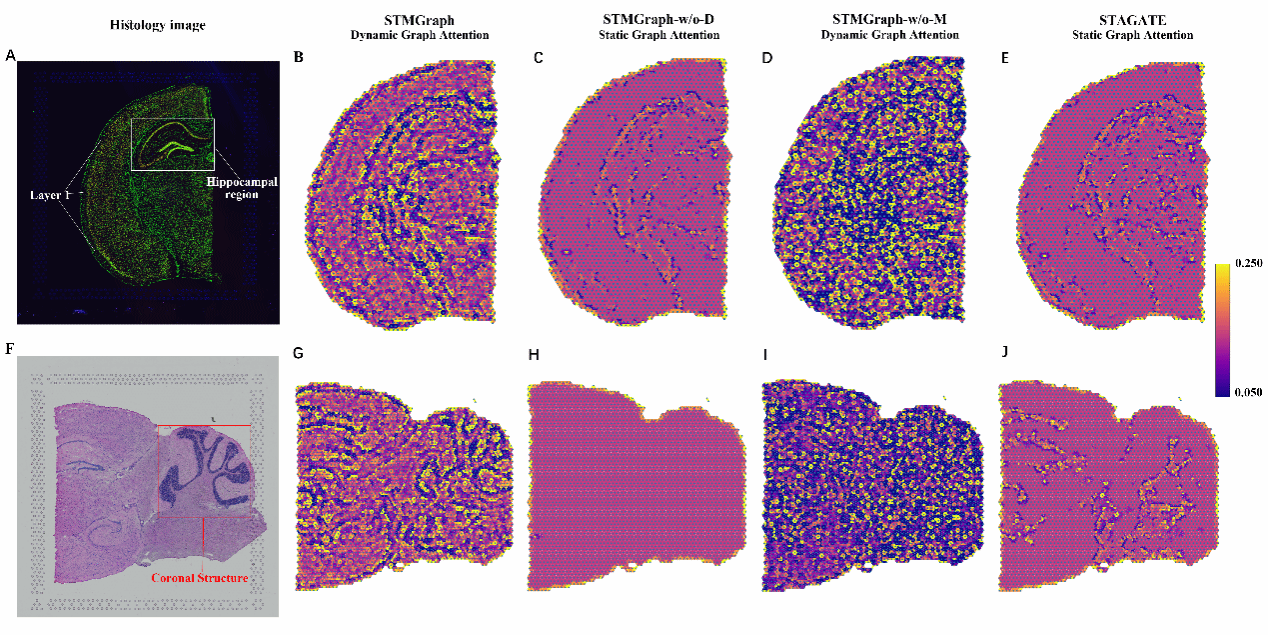
图2 STMGraph在10X Visium数据集的组织微环境中表现出优越的空间异质相似性的检测能力
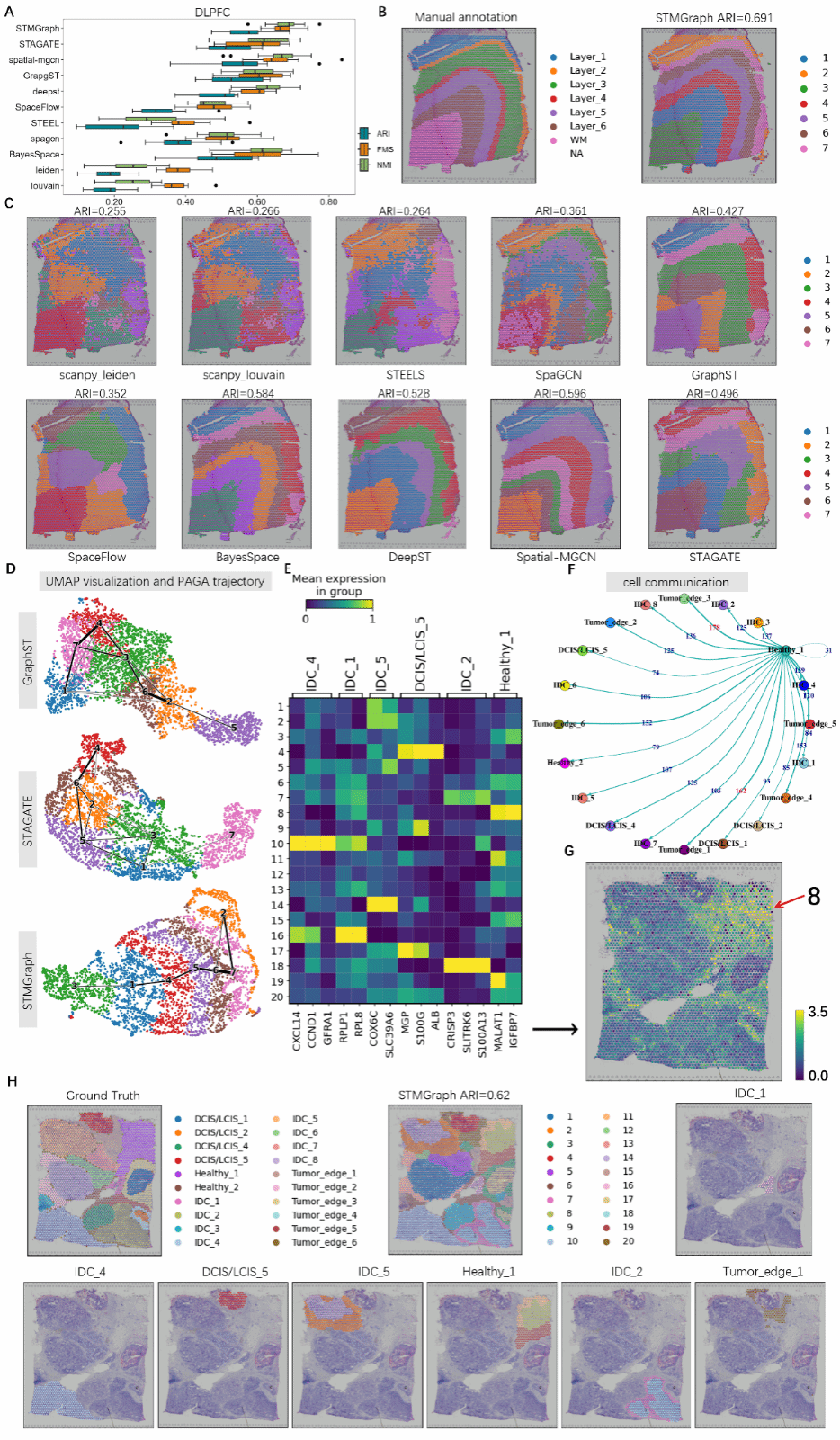
图3 基于10× Visium ST平台的STMGraph与10种最先进工具的基准测试
随后研究团队对10× Visium等多组数据进行了系统的基准测试,特别是在DLPFC(人类背外侧前额叶皮质)数据集上,STMGraph取得了卓越的性能表现。具体来说,STMGraph在该数据集上获得了最高的ARI=0.577、FMS=0.664和NMI=0.689的中位数评分。同样,STMGraph在小鼠前脑和人类乳腺癌数据中均能有较好的聚类表现。这些结果充分证明了STMGraph在空间域识别方面的优异性能(图 3)。得益于STMGraph优异的聚类表现,研究团队对人类乳腺癌数据进行细胞通讯分析,发现健康区域已经受到了旁侧癌细胞的侵袭,并且STMGraph能根据侵袭程度的不同将健康区域一分为二,远离癌细胞的8区域侵袭程度低,11和 19区域则容易受到癌细胞的作用。
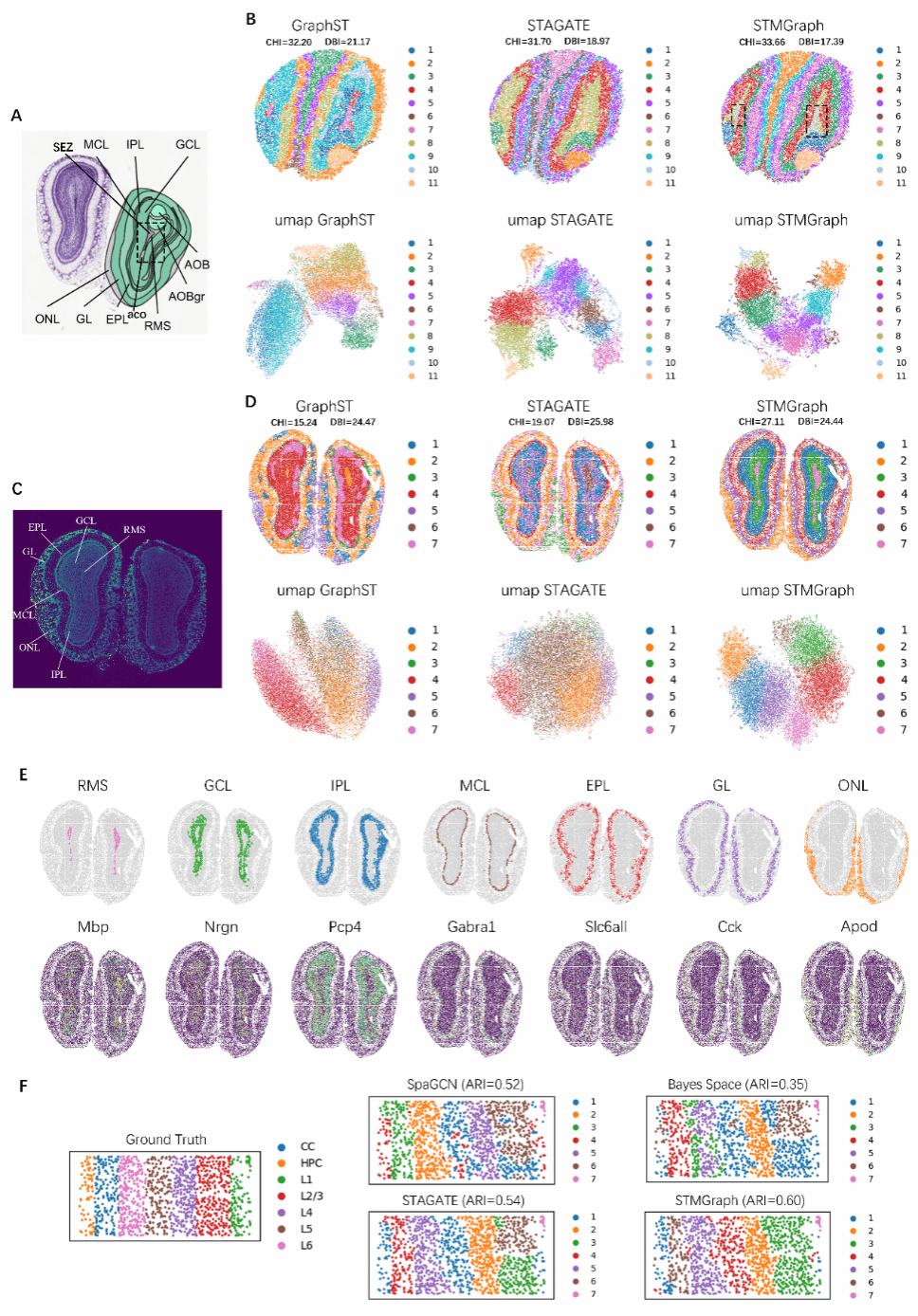
图4 STMGraph处理Stereo-seq、Slide-seqV2平台MOB和STARmap数据集时的优异性能
为了进一步验证STMGraph的广泛适用性,研究团队还测试了其他空间分辨率平台的数据集,包括Stereo-seq、Slide-seqV2和STARmap。特别是在小鼠嗅球组织中,STMGraph聚类结果展示为较大的CHI以及较小的DBI证明其类间差异大,簇内更紧密。在这些不同分辨率的数据集上,STMGraph依然展现出了最佳的聚类评估表现和强大的鲁棒性。这表明STMGraph不仅在低分辨率数据上表现优异,也能在高分辨率数据中保持高水平的性能(图4)。STMGraph优异的聚类性能也反映了DGAT结合掩码算法在节点、链路和图预测方面的优势,采用DGAT框架的STMGraph有效地提高了潜在嵌入的质量,最终使其对聚类的促进作用更加显著。总体而言,STMGraph在空间组学平台上对不同数据集的空间域识别方面表现出了卓越的能力,因此为寻找具有生物学意义的候选基因提供了潜在的预测能力,用于下游验证和肿瘤研究中的应用。
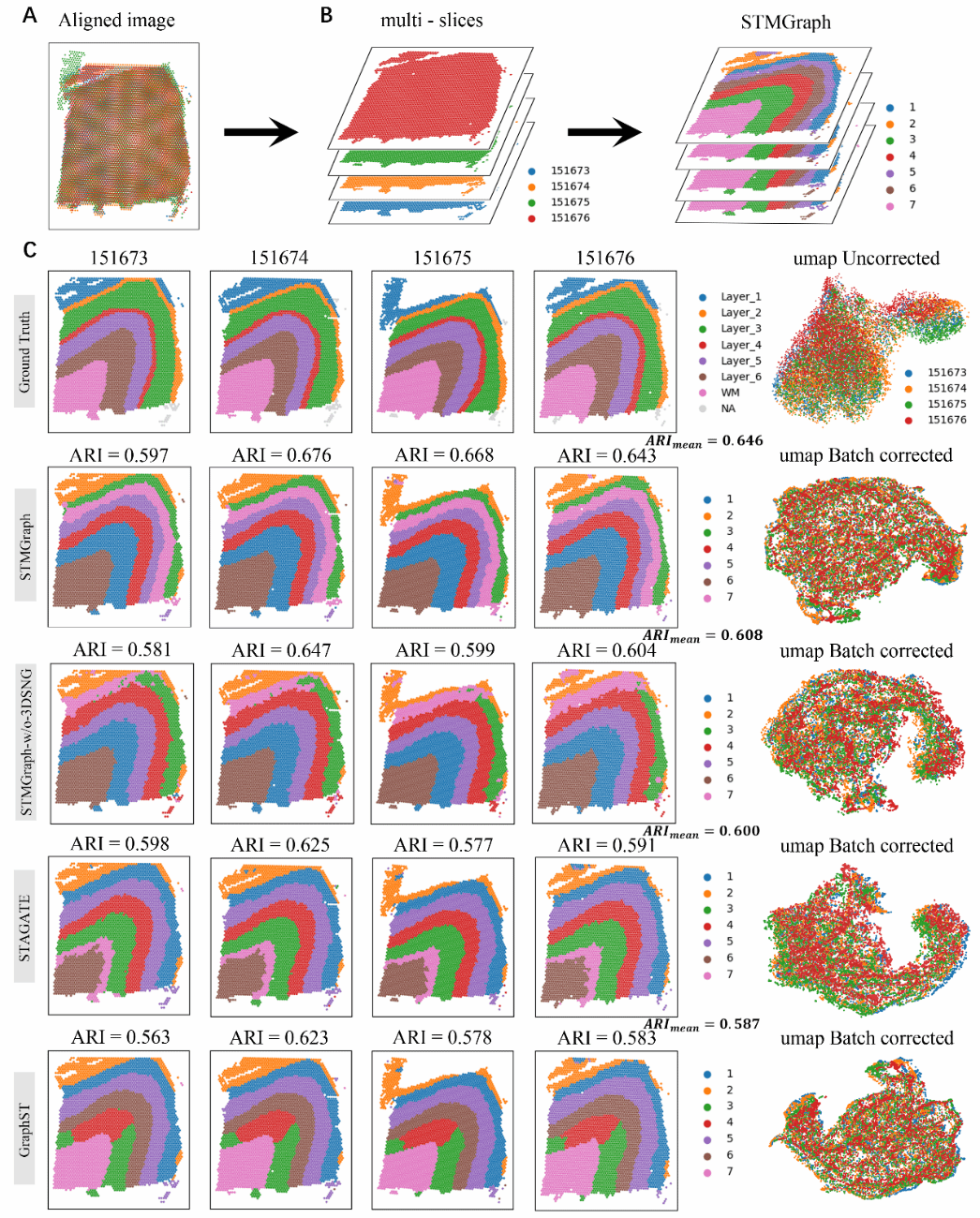
图5 DLPFC数据集(151673-151676)多片对齐后的空间域识别
STMGraph 融合了批量效应校正功能,专门用于处理垂直切片拼接的空间转录组数据聚类。这一功能在小鼠乳腺癌数据集上得到了显著验证,其中 iLISI(integration local inverse Simpson’s Index)评估值接近2,表明 STMGraph 具有强大的批次校正能力。此外,不同切片在 UMAP 可视化中能够均匀混合,进一步证明了 STMGraph 在减少批次效应方面的有效性。在处理平面对齐相接的小鼠脑区切片数据时,STMGraph 表现出卓越的性能,是唯一能够准确区分小鼠脑部冠状层的算法。为了进一步说明 STMGraph 的隐式批次校正对空间聚类的积极影响,研究团队使用 PASTE 对 DLPFC 四切片进行了垂直拼接对齐。处理后的 UMAP 图展示了不同切片能够均匀混合,且对应的ARI均值为最高的0.646(图5)。这一结果 不仅证明了 STMGraph 在多切片联合聚类中的优越性,还展示了其在处理大规模数据时的稳健性。STMGraph是一个不仅仅考虑本地SNG的spot的算法,还会聚合少量非本地SNG的spot,进而增强了其批次矫正能力。这种跨区域的信息整合显著增强了 STMGraph 的批次校正能力,使其在处理多切片数据时能够更有效地校正批次效应,提高聚类结果的准确性和可靠性。
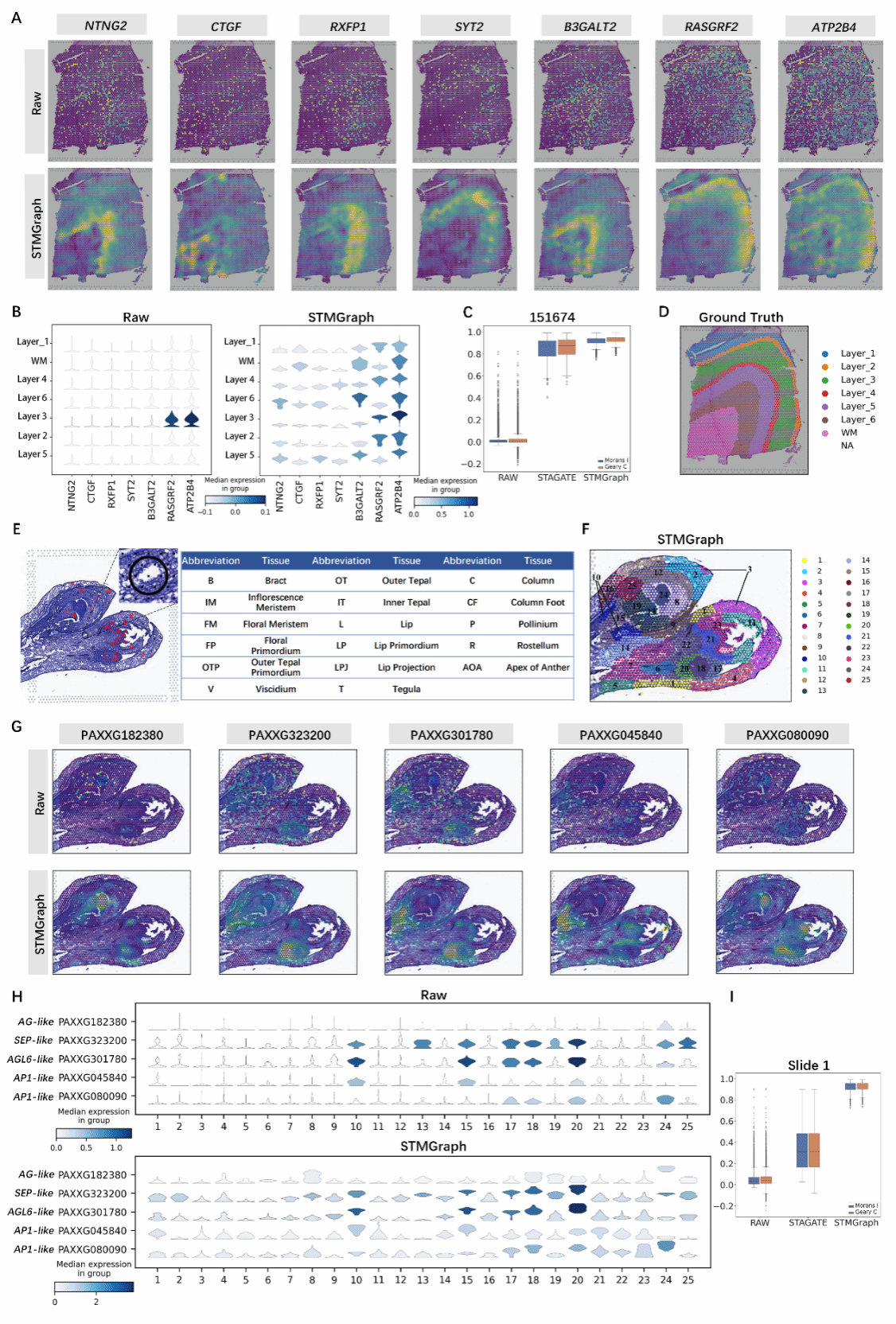
图6 STMGraph推断基因空间表达模式。
STMGraph显著提升DLPFC切片中的标记基因及蝴蝶兰(Phalaenopsis)花芽切片中MADs-box基因在不同花器官中的表达模式
最后,为了验证STMGraph对基因表达谱降噪的能力,研究团队选用了DLPFC数据集中#151674切片以及蝴蝶兰(Phalaenopsis)花芽切片进行实验。两组切片的原始空间转录组(ST)数据基因表达模式均较为混乱,基因与空间位置关联性弱。经过STMGraph降噪后,DLPFC切片中的标记基因以及蝴蝶兰花芽中的MADs-box基因的表达模式在相应的组织位置上变得更加清晰,空间相关性显著提升(图 6)。此外,降噪后的基因在Moran's I和Geary's C指数上更高,均优于原始基因以及经STAGATE处理后的基因,这充分证明了STMGraph在基因降噪方面的优越表现。STMGraph可以有效平滑基因表达的噪声,增强空间基因表达模式,降低原始数据中的表达谱无序性。
与动物组织切片相比,植物组织切片间隙多且大,导致空间位置的表达噪声高。团队利用蝴蝶兰花芽切片的空间转录组数据集测试STMGraph在植物组织中的降噪能力(图6E-I)。通过STMGraph降噪处理后,蝴蝶兰MADS-box基因在正确花器官位置中的表达显著增强,错误位置表达减弱。例如,PAXXG080090和PAXXG045840(AP1-like family)在花原基和分生组织中表达显著增强;PAXXG301780(AGL6-like family)在花瓣、蕊柱等组织中表达模式得到增强;PAXXG323200(SEP-like family)在花粉块中表达降低,而在蕊柱表达保持高水平;PAXXG182380(AG-like family)在蕊柱或雄蕊中表达增强,空间相关性显著提升。相比与原始数据和STAGATE降噪结果,STMGraph在蝴蝶兰花芽切片上基因空间位置表达降噪的得分(Moran's I和Geary's C指数)最高(图6I)。
综上所述,本研究开发了一个通用的空间转录组学深度学习框架STMGraph,它将双重掩码(MASK-REMASK)与动态图注意模型(DGAT)相结合,充分考虑细胞的空间依赖性,通过空间上下文感知将局部和非局部特征结合起来。文章有四大亮点1. 提出一种不完全依赖空间转录组原始测序数据的高精准度空间域识别算法。2. 通过提高网络的链路预测与图预测能力,提出一种不依赖预聚类的微环境异质相似性检测方法。3. 极大的提高了算法去批次性能,实现了对更大组织面积以及垂直结构的空间组织分析,为未来处理更大数据量的组学分析奠定了基础。4. 实现了基因降噪,增强基因在空间域的表达能力。同时算法能适应不同分辨率的空间转录组平台(multi- to sub-cellular resolutions)。
福建农林大学海峡联合研究院基因组与生物技术研究中心与生命科学学院已毕业研究生林礼贤为第一作者;福建农林大学海峡联合研究院基因组学与生物技术研究中心马晓开教授为论文通讯作者。此外,参与本研究的还有福建农林大学海峡联合研究院基因组与生物技术研究中心研究生王浩宇、陈煜骁、王媛媛、许玉洁、陈政霖、杨跃敏、刘锟鹏。福建农林大学海峡联合研究院为第一单位和通讯作者单位。该研究得到了国家自然科学基金面上和青年项目以及福建省自然科学基金面上项目的资助。
原文地址:https://doi.org/10.1093/bib/bbae685

