近日,福建农林大学基因组与生物技术研究中心发表了题为“Assembly of allele-aware, chromosomal-scale autopolyploid genomes based on Hi-C data”的研究论文,该项研究成果于2019年8月5日在线发表于国际著名学术杂志《自然·植物学》,福建农林大学为文章的第一作者单位和通讯作者单位。基因组中心张兴坦副教授为论文第一作者。基因组中心科研助理张晟铖,植物保护学院赵茜博士参与了该项研究工作。
同源多倍体指的是同一物种经过染色体加倍形成的多倍体,在植物界比较常见,农业育种上有重要应用价值。然而,同源多倍体基因组的组装却是世界性难题。虽然近期发展的染色质构象捕获(Hi-C)技术为染色体水平的组装提供了便利,但组装同源多倍体或者近期加倍的异源多倍体时,这一技术仍然面临着很大的挑战。目前除甘蔗基因组以外,尚没有其他同源多倍体基因组可以准确组装到染色体水平,这是由于多倍体基因组内多个单倍型之间存在很高的序列相似性。当用Hi-C技术挂载染色体时,同源染色体之间的Hi-C交联信号会将序列相似度极高的等位片段连接在一起,导致同源染色体被错误地连接到一起,形成大量嵌合的组装。
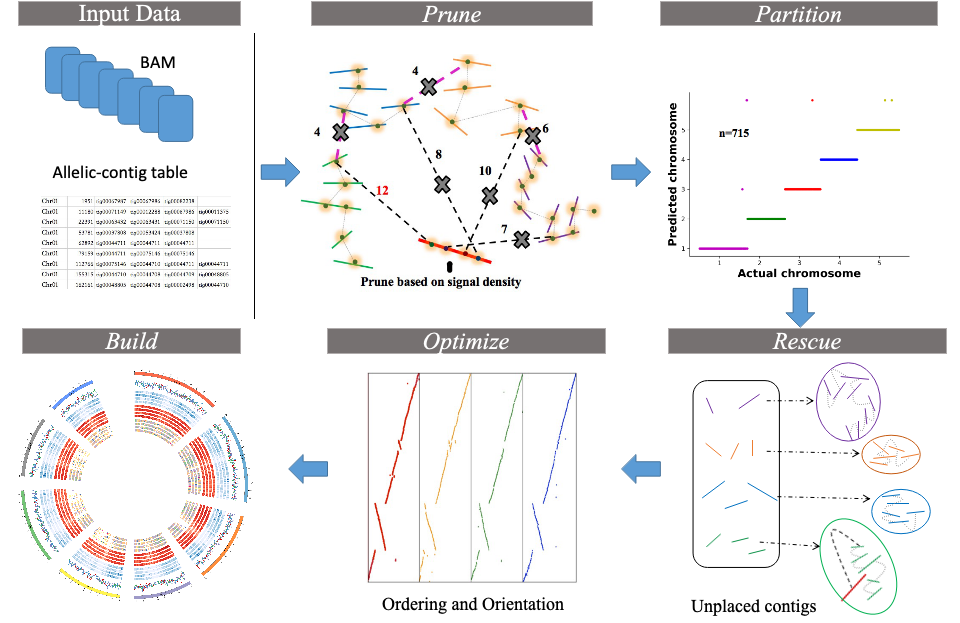
图 ALLHiC算法流程和功能模块
此次发表的论文公布了ALLHiC算法,通过修剪同源染色体之间的交联信号,将等位基因和同源序列分隔在各自的单倍型内独立组装,从而减少了大量拼接错误。这个方法学上的突破使得一系列重要多倍体基因组直接从头组装成为可能,比如苜蓿、土豆等,或可用于修复已公布的多倍体物种基因组组装序列中的错误。
该研究团队多年来一直致力于复杂基因组和多倍体基因组学研究,并于2018年8月份利用ALLHiC算法成功破译同源多倍体甘蔗割手密基因组,相关成果已于当年发表在《自然·遗传学》期刊上。该团队于2015年提出了ALLMAPS算法整合多张基因组图谱,提高组装的准确性和完整性,迄今引用100余次,并用于破译高度杂合的菠萝基因组,相关成果发表在当年的《自然·遗传学》期刊上。
文章链接:https://www.nature.com/articles/s41477-019-0487-8

